1、概述
2015年提出的用于生物图像分割的网络,并在当时取得了冠军- [实际也可用于其他领域,比如一些
Kaggle图像分割比赛中也有使用,并且取得了很好的成绩]
- [实际也可用于其他领域,比如一些
- 一般的
CNN是预测一张图片的类别,而图像分割需要预测每个像素属于的类别,使用是全卷积网络FCN(fully convolutional network) - 网络结构包括收缩 [就是
CNN的卷积操作]和对应的扩展 [可以进行反卷积,这里采用上采样(upsample)]- 收缩主要用于捕捉上下文特征
- 扩展用于定位
- 需要很少的训练集即可完成训练,很容易收敛
2、网络结构

- 左半部分收缩就是平常的
CNN卷积操作- 经过卷积、
Relu和2x2 max pooling
- 经过卷积、
右半部分扩展采用的上采样(
upsampling)操作,同时拼接上对应的左半部分的feature maps- 因为前面几层的卷积层分辨率比较高,定位比较准确
- 后面的几层卷积层分辨率比较低,分类比较准确
- 所以把低分辨率和高分辨率的
feature map拼接起来,得到更好的结果
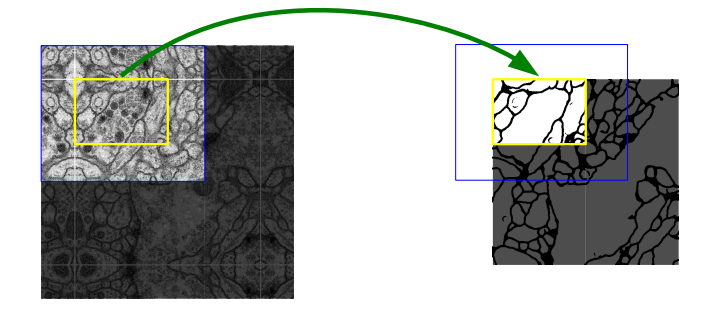
如果预测时输入图片大小有问题,可以使用镜像拼接方式,同时也可以调整输入的大小是偶数,方便进行
2x2 max pooling- 图中黄色部分待预测,需要蓝色部分作为输入,对称的方式生成周围的部分。
3、训练
- 损失函数使用逐像素的
softmax函数和交叉熵损失函数的结合 Softmax函数:$${p_k(x)} = {e^{a_k(x)} \over \sum_{k’=1}^K e^{a_{k’(x)}}}$$- $a_k(x)$表示在
feature maps中的的channel=k的feature map像素位置为x的激活值 - $K$是类别数
- [就是单个
feature map上每个像素的类别概率]
- $a_k(x)$表示在
- 训练需要标注对应的
mask,就是类别的区域标记
- 因为使用了
Relu激励函数,对应的权重初始化方法为标准差为$\sqrt{2/N}$的高斯分布,具体关于不同激励函数对应的输出花方法可以看这里 - 训练集小的话可以做数据增强
4、Keras 中的实现
- 借鉴
github上实现好的:点击查看,版本:Keras (2.0.8),tensorflow (1.3.0) 两层卷积操作函数
- 判断是使用
theano还是tensorflow作为backend, 因为他们对应的数据维度不同 - 可以使用
BN和Dropout操作 - 两层卷积也就对应了上面
U-net结构图的两个卷积操作12345678910111213141516def double_conv_layer(x, size, dropout, batch_norm):if K.image_dim_ordering() == 'th':axis = 1else:axis = 3conv = Conv2D(size, (3, 3), padding='same')(x)if batch_norm is True:conv = BatchNormalization(axis=axis)(conv)conv = Activation('relu')(conv)conv = Conv2D(size, (3, 3), padding='same')(conv)if batch_norm is True:conv = BatchNormalization(axis=axis)(conv)conv = Activation('relu')(conv)if dropout > 0:conv = Dropout(dropout)(conv)return conv
- 判断是使用
构建网络
- 最后是
1x1的卷积,使用的Sigmoid函数作为最后的输出概率123456789101112131415161718192021222324252627282930313233343536373839404142434445464748def ZF_UNET_224(dropout_val=0.0, batch_norm=True):print("con")if K.image_dim_ordering() == 'th':inputs = Input((INPUT_CHANNELS, 224, 224))axis = 1else:inputs = Input((224, 224, INPUT_CHANNELS))axis = 3filters = 32conv_224 = double_conv_layer(inputs, filters, dropout_val, batch_norm)pool_112 = MaxPooling2D(pool_size=(2, 2))(conv_224)conv_112 = double_conv_layer(pool_112, 2*filters, dropout_val, batch_norm)pool_56 = MaxPooling2D(pool_size=(2, 2))(conv_112)conv_56 = double_conv_layer(pool_56, 4*filters, dropout_val, batch_norm)pool_28 = MaxPooling2D(pool_size=(2, 2))(conv_56)conv_28 = double_conv_layer(pool_28, 8*filters, dropout_val, batch_norm)pool_14 = MaxPooling2D(pool_size=(2, 2))(conv_28)conv_14 = double_conv_layer(pool_14, 16*filters, dropout_val, batch_norm)pool_7 = MaxPooling2D(pool_size=(2, 2))(conv_14)conv_7 = double_conv_layer(pool_7, 32*filters, dropout_val, batch_norm)up_14 = concatenate([UpSampling2D(size=(2, 2))(conv_7), conv_14], axis=axis)up_conv_14 = double_conv_layer(up_14, 16*filters, dropout_val, batch_norm)up_28 = concatenate([UpSampling2D(size=(2, 2))(up_conv_14), conv_28], axis=axis)up_conv_28 = double_conv_layer(up_28, 8*filters, dropout_val, batch_norm)up_56 = concatenate([UpSampling2D(size=(2, 2))(up_conv_28), conv_56], axis=axis)up_conv_56 = double_conv_layer(up_56, 4*filters, dropout_val, batch_norm)up_112 = concatenate([UpSampling2D(size=(2, 2))(up_conv_56), conv_112], axis=axis)up_conv_112 = double_conv_layer(up_112, 2*filters, dropout_val, batch_norm)up_224 = concatenate([UpSampling2D(size=(2, 2))(up_conv_112), conv_224], axis=axis)up_conv_224 = double_conv_layer(up_224, filters, 0, batch_norm)conv_final = Conv2D(OUTPUT_MASK_CHANNELS, (1, 1))(up_conv_224)conv_final = BatchNormalization(axis=axis)(conv_final)conv_final = Activation('sigmoid')(conv_final)model = Model(inputs, conv_final, name="ZF_UNET_224")return model
- 最后是
5、总结
- 图像分割能够获得很好的结果,要求的训练集比较小
- 包括了收缩和扩展两部分,扩展部分拼接了对应的收缩部分的
feature maps
Reference
- https://arxiv.org/abs/1505.04597#
- https://lmb.informatik.uni-freiburg.de/people/ronneber/u-net/
- https://github.com/ZFTurbo/ZF_UNET_224_Pretrained_Model
- https://github.com/jakeret/tf_unet
- 本文链接: http://lawlite.me/2017/10/18/论文记录-U-Net-Convolutional-Networks-for-Biomedical-Image-Segmentation/
-
版权声明:
本博客所有文章除特别声明外,均采用 CC BY-NC-SA 3.0 许可协议
 。转载请注明出处!
。转载请注明出处!

